0) 의료데이터에서 기존 PCA와 같은 차원 축소 모델을 사용하지 않는 이유
- PCA(주성분 분석)의 경우 선형 차원축소 방식으로, 의료 데이터와 같이 데이터가 교차되며 비선형 구조를 가지는 데이터의 차원을 축소하기에 적합하지 않음.
- 이에, * 역 그래프 임베딩을 기반으로 한 기본 그래프 구조의 로컬 정보를 캡쳐하는 아래와 같은 모델이 주로 사용됨
- * 역 그래프 임베딩 : 기본 그래프 로컬 정보 구조(한마디로, 본래의 형태..?) 를 캡처하는 기법(모델)
1) DDRTree
| 구분 | Desc |
| 정의 | 고차원 공간의 데이터 포인트를 저차원의 |
| 장점 |
|
| 단점 |
|
| Parameter |
|
| 구현결과 |
+) SimplePPT
 |
2) UMAP
| 구분 | Desc |
| 논문 | UMAP: Uniform Manifold Approximation and Projection for Dimension Reduction |
| github | https://github.com/lmcinnes/umap (CPU, scikit-learn, Tensorflow 기반, 다만 매우빠르다고함 → 아직 미확인)
|
| 정의 |
|
| 장점 |
|
| 단점 | |
| 기타 |
|
| 구현결과 |
(+) SimplePPT
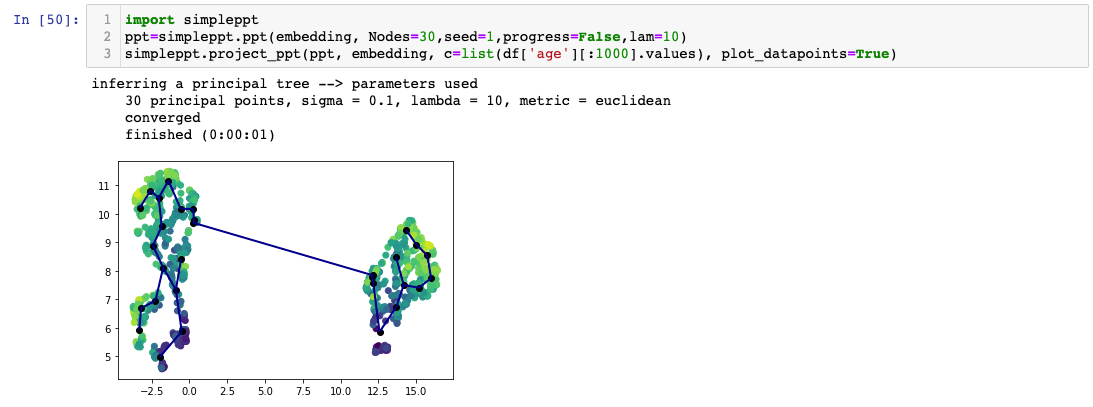 |
3) DDRTree vs UMAP 비교 테스트 결과
- 조건
- 스코틀랜드 당뇨데이터 sample 1000명 데이터로 비교
- 구심점의 개수(parameter)는 10개로 동일하게 설정
- 비교결과
- 속도
- UMAP(6.07s) >> DDRTree(19.39s)
- 표현형 시각화 비교 (UMAP > DDRTree)
- 스펙트럼에서 각 표현형을 시각화 했을때, DDRTree의 경우 표현형의 수치별 환자가 분산되어있으나, UMAP의 경우 비교적 밀집되어있음
- 당뇨여부 시각화 비교 (UMAP >> DDRTree)
- DDRTree에 비하여 UMAP이 스펙트럼의 특정구간에 당뇨환자를 확연히 밀집하여 표현하고 있음.
- 속도

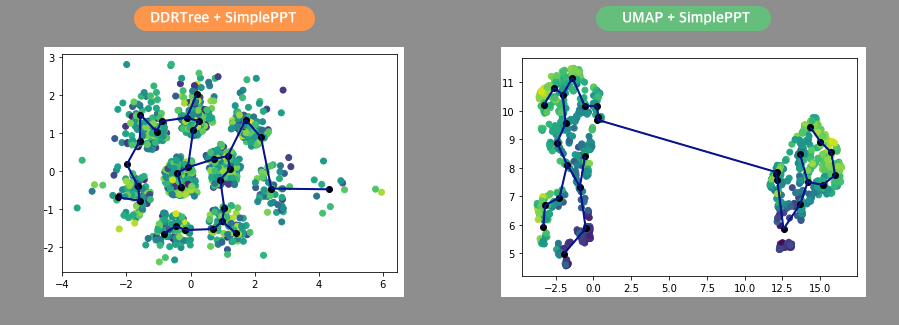
추가) SimplePPT → 차원축소모델과 함께 활용 (본래 차원의 그래프 구조 학습을 통해 도출한 주요 포인트를 이어 트리를 시각화..)
| 구분 | Desc |
| 논문 | Ref . Mao et al. (2015), SimplePPT: A simple principal tree algorithm, SIAM International Conference on Data Mining.
|
| github | https://github.com/LouisFaure/simpleppt (GPU를 통한 가속 가능) |
| 정의 |
|
| 장점 |
|
| 단점 |
'DEVELOP_NOTE > ML' 카테고리의 다른 글
| 컨텐츠 기반 추천 vs 협업 필터링 (0) | 2023.06.26 |
|---|---|
| [Real-time Instance Segmentation] 화상대화 배경제거 (1) (0) | 2023.01.09 |
| [Meta Learning] Few-Shot Learning을 알아보자! (2) | 2022.09.29 |
| [VGG]VGGNet을 한번 파헤쳐보자! (0) | 2022.09.17 |
| [CNN]CNN(Convolutional Neural Networks) : 합성곱 신경망을 파헤쳐보자! (0) | 2022.09.07 |



